Séquençage de plasmides
Séquençage de plasmide
En utilisant une méthode de séquençage de troisième génération (Oxford Nanopore Technologies (ONT)), notre service permet un séquençage de haute qualité de l’entièreté de vos plasmides à un prix abordable ($15 CAD/plasmide).
Formulaire disponible ici.

Ce service s’adresse à vous si:
- Vous avez un long insert à séquencer (qui nécessite plusieurs réactions de séquençage de type Sanger).
- Vous possédez ou vous avez reçu un plasmide dont vous ne connaissez pas la séquence.
- Vous voulez vérifier qu’un plasmide que vous utilisez depuis longtemps ne possède pas de mutation inattendue dans le squelette de celui-ci.
Ce service est peut-être moins adapté à vous si:
- Vous ne vous intéressez qu’à connaitre la séquence d’un petit insert d’un plasmide.
Nous effectuons désormais un séquençage chaque jeudi, garantissant un délai de traitement plus prévisible. Les échantillons reçus au plus tard le mercredi seront inclus dans le prochain séquençage. Contactez-nous si vous avez des questions sur la planification.
- Ne nécessite pas d’amorces spécifiques.
- Permet la détection d’espèces aberrantes dans votre préparation de plasmide (tel que les dimères, trimères, etc.).
- Vous donne à tous les coups la séquence du squelette de votre plasmide.
- Ne produit pas de régions de mauvaise qualité (telles que le début et la fin de réactions de séquençage de type Sanger).
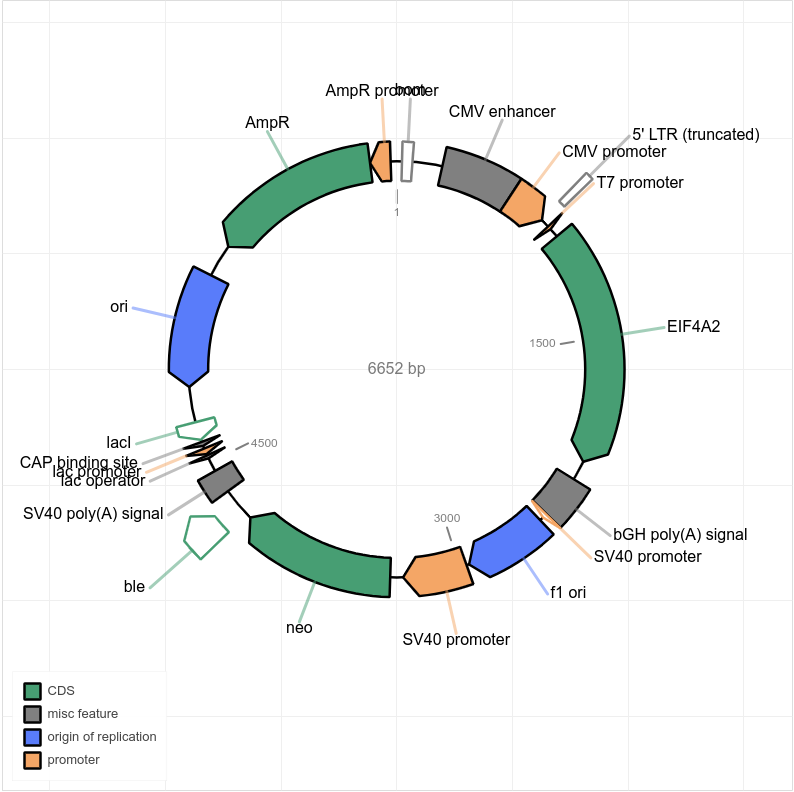
De plus, notre service vous donne aussi en prime une carte annotée de votre plasmide (par l’utilisation de l’excellent outil pLannotate).
- 20 µL de plasmide à 40 ng/µL. *Le plasmide doit avoir été purifié sur colonne à l'aide d'un kit commercial. Si la purification a été effectuée avec un protocole maison, les résultats du séquençage peuvent être de moindre qualité ou échouer complètement.
- Du formulaire complété contenant quelques informations sur vos échantillons (dont vous pouvez télécharger le gabarit ici).
- La séquence de référence (facultatif). Si celle-ci est fournie, elle ne servira qu’à aligner la séquence consensus produite à votre séquence de référence ainsi qu’à adapter (rotation et/ou complément inverse) la séquence consensus, ce qui vous facilitera la tâche lors de l’analyse.
- Une séquence consensus (.fasta).
- Un fichier relatant les statistiques de chaque base de la séquence consensus (.xlsx).
- Deux graphiques rapportant la qualité ainsi que la taille des lectures observées lors du séquençage (.png).
- Une carte annotée de la séquence consensus pouvant être visualisée dans un navigateur web (.html) ou avec l’aide d’un logiciel tel que snapgene ou snapgene-viewer (.gbk).
- Un alignement de la séquence consensus avec votre séquence de référence (.aln, si vous avez fourni celle-ci). Cet alignement peut être visualisé de façon pratique à l’aide du logiciel snapgene ou snapgene-viewer.
